【RDKit】化合物の共鳴構造を列挙する
RDKitで化合物を扱う際に、共鳴構造の列挙をする必要がでてくる時があるかと思います。
その方法を調べてみたところ、その方法について解説している記事がなかったため、この記事で簡単にまとめることにしました。
環境・ライブラリ
- Python: 3.6
- RDKit: 2020.03.3.0
やり方だけ知りたい人用
解説とかいらないからとりあえず方法だけ知りたいって人は以下のスクリプトを参考にして下さい。
1 | from rdkit import Chem |
共鳴構造を持つ化合物の用意
今回はとりあえず3つの共鳴構造を持つナフタレンに対して共鳴構造を列挙してみたいと思います。
1 | from rdkit import Chem |
ResonanceMolSupplier を使って共鳴構造の列挙
RDKitで化合物の共鳴構造を列挙するためには rdkit.Chem.ResonanceMolSupplier class を使います。
他のSupplier class と使い方は同じです。
1 | from rdkit.Chem import ResonanceMolSupplier, ResonanceFlags |
#0: C1=CC2=C(C=C1)C=CC=C2 |
描画すると以下のような感じです。
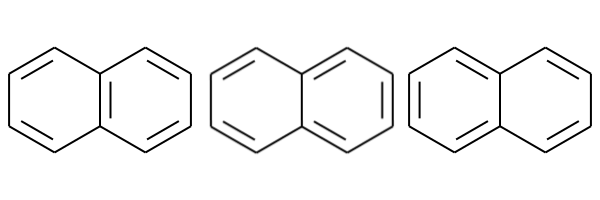
正しく共鳴構造が列挙できていることが確認できました。
単純に共鳴構造の数が欲しいだけの場合は、len(suppl) で取得できます。
ResonanceFlags の種類
今回は全ての共鳴構造を列挙したかったため、 ResonanceFlags.KEKULE_ALL をオプションとして指定しました。
他には以下のオプションがあるため、必要に応じて指定して下さい。
- ALLOW_CHARGE_SEPARATION
- ALLOW_INCOMPLETE_OCTETS
- UNCONSTRAINED_ANIONS
- UNCONSTRAINED_CATIONS
終わりに
あまり使う頻度としては高くないかと思いますが、RDKitで共鳴構造を列挙したい時は今回紹介した方法を使うと簡単にできます。
大量に共鳴構造が得られることがわかっている場合は、SetNumThreads メソッドでスレッド数を指定すれば並列で計算できるので時短できます。
また列挙する最大の構造数は1000になっているので、必要に応じてmaxStructs で指定して下さい。
それでは
参照
【RDKit】化合物の共鳴構造を列挙する
https://sishida21.github.io/2020/07/31/rdkit-enumerate-resonance-structures/