【RDKit】SAscoreの計算時に注意するポイント
分子の合成容易性を見積もる際には、一般的にSAscoreという指標を用いることが多いです。
RDKitではSAscoreを簡単に計算することが可能なので多くの方が利用しているかと思いますが、これを計算する際に認識しておくべき注意点に遭遇しました。
そこで本記事ではSAscoreを計算する際の注意点についてまとめようと思います。
SAscoreについて
SAscoreを同計算しているかが気になる方は、直接rdkit/Contrib/SA_Score/sascorer.pyを参照してください。
SAscoreそのものを勉強したい方は、原著論文もしくはこちらのブログを参照してください。
以下に本記事を理解する上での最低限のSAscoreに関する情報を記載しておきます。
- SAscoreは1~10の範囲で算出され、1(易) ~ 10(難)と解釈する
- SAscoreの計算式にはペナルティ項(難寄りのスコアになる)が含まれている
- ペナルティ項の一つにサイズペナルティ(大きい分子はスコアを高くする)がある
それでは本文に入ります。
環境・ライブラリ
- Python: 3.8
- rdkit: 2021.03.4
- jupyter: 1.0.0
背景
ある分子のSAScoreを計算した際に、今までの感覚では2~3くらいのSAscoreになりそうな分子が6くらいのスコアになっていて違和感を感じた。
原因
SAscoreを計算する際に入力する対象分子のMolオブジェクトに水素を明示的に付加していたことが原因だった。
以下にアスピリンを例に明示的な水素付加あり/なしでのSAscoreの違いを実際に示す(Jupyter Notebookで実行してください)。
1 | import os |
1 | smi = "CC(=O)OC1=CC=CC=C1C(O)=O" # aspirin |
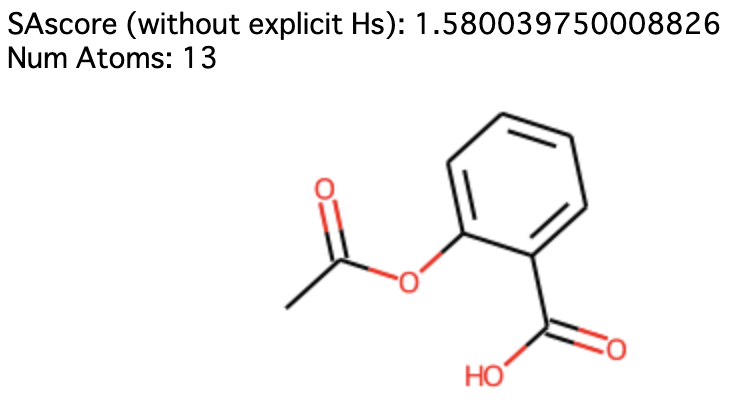
1 | smi = "CC(=O)OC1=CC=CC=C1C(O)=O" # aspirin |
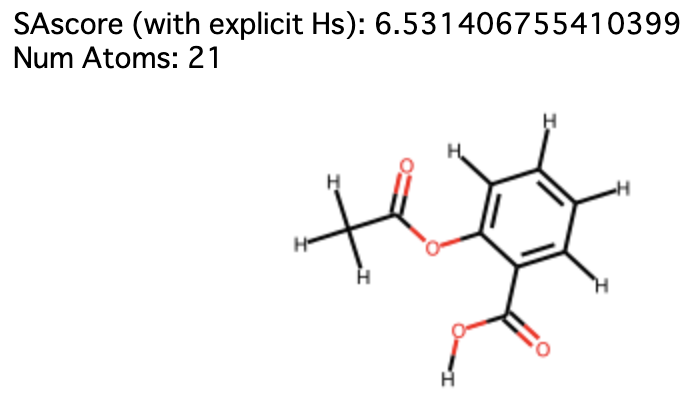
解説
このような現象がなぜ起きてしまうかというと、 Chem.AddHs() で分子に水素を付加すると Chem.Mol.GetNumAtoms() で原子数を計算する際に付加した水素の数まで含まれてしまうのが起因している。
RDKitのMolオブジェクトでは、通常は原子数のカウントに水素の数は含まれていない。
そしてSAscoreのペナルティ項の一つであるサイズペナルティはこの原子数をもとに算出しているため、原子数に水素の数まで含まれていると大きい分子と判定されてSAscoreが高く算出されてしまっていたのがことの顛末でした。
おわりに
本記事ではSAscoreの計算時の注意点についてまとめました。
今回のようなケースはエラーとしてでることはないのでなかなか気づきにくいと思います。
水素付加の有無でRDkitの動作が影響受ける可能性があることを認識しておくことで意図しない計算ミスを避けることができますので、今回のようなケースを頭の片隅に置いておくのがよいかなと思います。
それでは。
【RDKit】SAscoreの計算時に注意するポイント
https://sishida21.github.io/2021/08/07/rdkit-sascore-calculation-caution/